随着快速、经济的微生物基因组测序技术的发展,大量的微生物基因组数据被公布。可以利用全基因组水平的基因扫描和等位基因信息等对菌种进行差异菌株分析。差异菌株分析在研究样本菌株在群体进化或分子流行病学方面的关系有广泛应用。AG亚游生物对细菌基因组测序提供以下多菌株差异比较分析服务。
结果展示
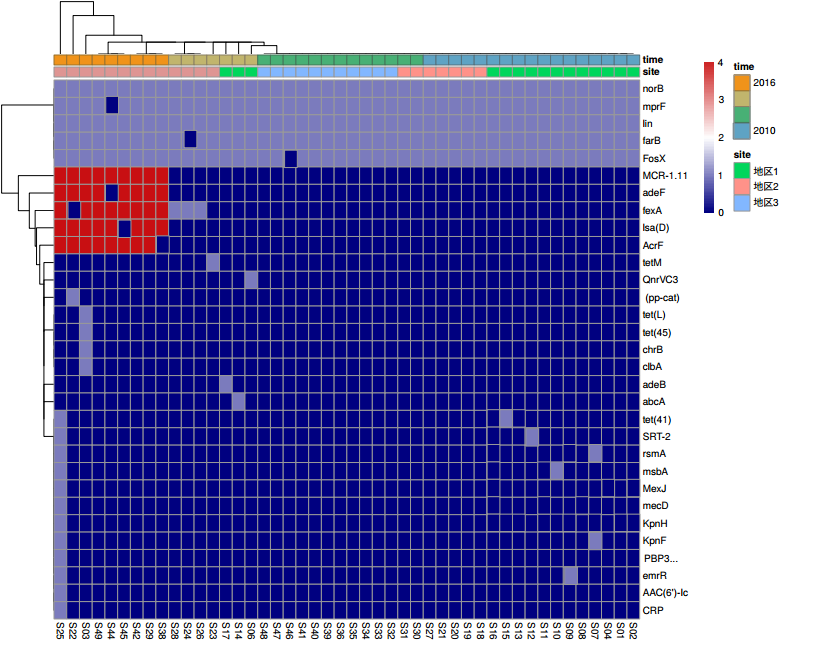
1) CARD 数据库比对分析
The Comprehensive Antibiotic Resistance Database ( http://arpcard.mcmaster.ca ) CARD 数据库核心是 ARO(Antibiotic Resistance Ontology) , ARO 包含了与抗生素抗性基因,抗性机制,抗生素和靶相关的 term 。 CARD 数据库是目前应用广泛的耐药基因研究工具。
抗性基因热图
2) VFDB 数据库比对分析
毒力因子数据库 VFDB ( http://www.mgc.ac.cn/VFs/main.htm )由中国医学科学院研发,被广泛应用于毒力因子基因鉴定。 该数据库目前已涵盖 74 个属,一千余个致病因子,三万多条基因序列。是目前公开的、细菌致病因子应用广泛的数据库。
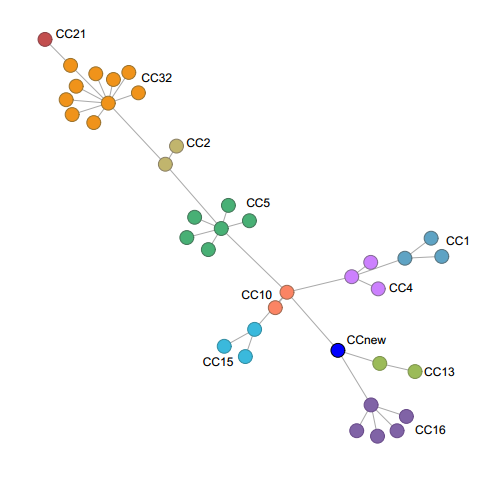
3) MLST 分析
MLST ( Multi-locus sequence typing )多位点序列分型是常用的基因分型方法。通过 PCR 扩增 6~10 个管家基因内部片段,测定其序列,得到等位基因编号,然后进行等位基因图谱 (allelic profile) 或序列类型 (sequence types , STs) 鉴定,再根据等位基因图谱使用配对差异矩阵 (matrix pair-wise differences) 等方法构建系统树图进行聚类分析。
MLST 最小生成树
4) cgMLST 分析
cgMLST (Core Genenome multi locus sequence typing) 核心基因组多位点序列分型是基于全基因组的核心靶基因进行序列分型,在现有的 MLST 数据基础上,以大量菌株中存在的核心基因组作为序列分型标记。 cgMLST 的标记位点多,密度高,其对菌株之间的细微变异也更为敏感,进一步提高了结果的精确度和准确性。将菌株分型鉴定的分辨率拓展到克隆级别。
细菌 cgMLST 分型有助于菌株进化研究,感染的早期发现、耐药菌株溯源和传播途径甄别,在菌株进化研究、流行病学溯源调查等方面均表现出。
cgMLST 最小生成树
5 )SNP分析
SNP 即 Single Nucleotide Polymorphism ,主要是指在基因组水平上由单个核苷酸的变异所引起的 DNA 序列多态性,包括单个碱基的转换、颠换等。使用核心基因的 SNP ( cgSNP )信息构建系统进化树,分辨率最高,但只适合特别近的菌株。
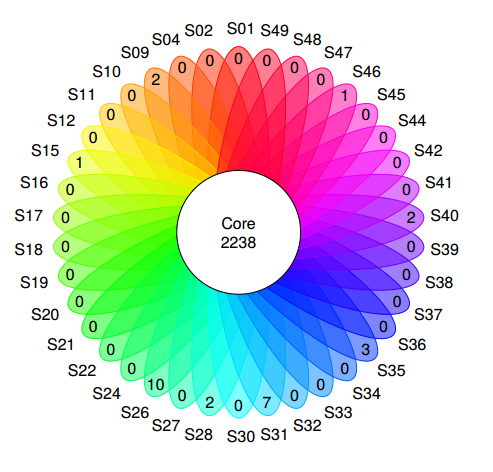
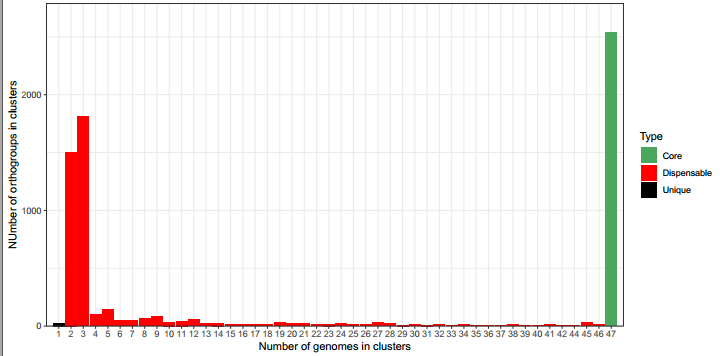
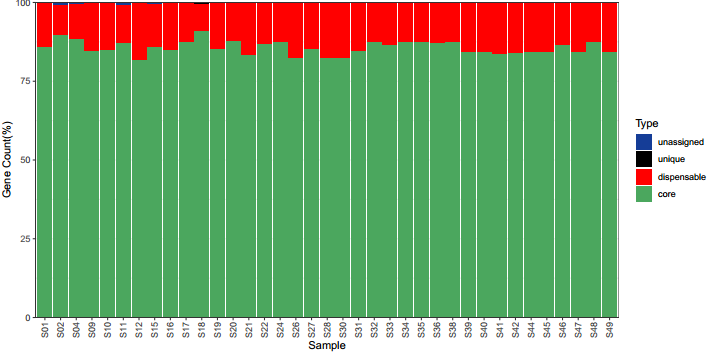
6) 泛基因分析
泛基因组即某一物种全部基因的总称。对各个样本中的不同类型的基因占比进行统计,从而对样本中基因的结构类型有宏观的把控。泛基因组分为核心基因组( core genome )、可变基因组( dispensable ),特有基因 ( unique genes )。 其中 unique gene 为样本特有基因,一般与该样本特有的表型、特有的适应性等区别于其他样本的特征相关; dispensable gene 为样本中的附属基因,这部分基因形成了群体的遗传多样性; core gene 为所有样本共有的基因,与群体共有或基本特征相关,形成了某类物种特有的表型或者 属性。
核心基因家族的统计
同源基因家族的分布统计
各样本同源基因的分布统计
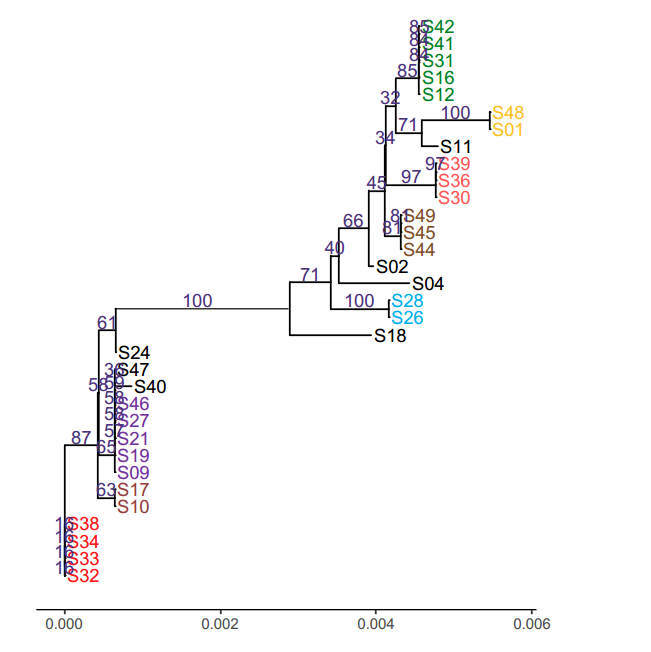
7) 系统进化分析
GTDB(GENOME TAXONOMY DATABASE)这一分类系统以细菌中普遍存在的120个单拷贝蛋白质(bac120)为基础;在对多分组类别消歧后,根据相对演化散度标准化和分级,得到基因组分类数据库(GTDB ),将目的序列和 GTDB 数据库比对,比对到的单拷贝蛋白序列进行 alignment ,而后进行发育树的绘制。

 AG亚游生物 您自己的微生态研究团队|专注微生态研究与应用
AG亚游生物 您自己的微生态研究团队|专注微生态研究与应用 




-1.jpg)
-2.jpg)
-3原始.jpg)

