细菌存在于 根尖周 炎 牙髓病灶的组织切片 中
格微 ® 甄选 菌种特异性 16S FISH探针设计服务
Ø 从 198 万条已知的全部 16S数据库 中,评测 目标微 生物全部可能的 FISH探针,报告出特异性最佳的一条,以及可能的错配(基于目前全部的 198 万条 16S序列)。
Ø 如单条 FISH探针,特异性不佳(很难精确到菌种),则评估双FISH探针(两条目标区域独立、不重合的1 6 S FISH探针)的特异性情况,给出详细的解读报告。
Ø 介于 2 3 S已知序列条数约2 2 . 7 万条,只有 1 6 S已知序列1 /10 的数量,AG亚游 FISH探针的设计都基于1 6 S序列开展。
AG亚游生物尽力为客户提供最佳的细菌 特异性探针为目标 ,利用当前较为完善的数据库系统及自身开发的程序完成对16S序列数据库全面 筛选匹配,协助科研客户挑选出特定菌种特异性最佳的探针 。 咨询电话: 021-50763698。
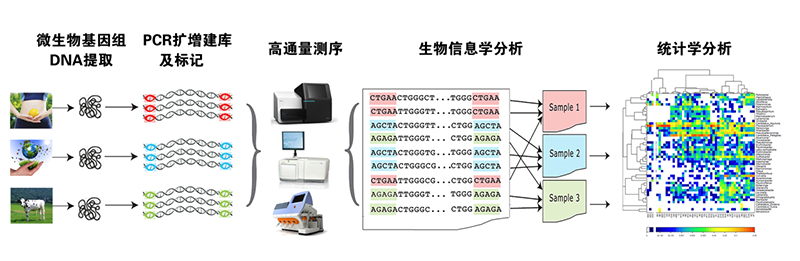
细菌 FISH检测 是以微生物中较稳定的 rRNA (主要是16S和23S) 作为分析的目标,以rRNA序列设计探针进行杂交,对待检测的微生物分布情况和特征性的微生物进行鉴定与定量分析,提供微生物形态学、空间分布和生物体的细胞数量等信息。

FISH杂交模式图 新型寡聚核苷酸荧光原位杂交:发展与应用 2023年
样本类型
粪便、土壤、水样、昆虫、植物根部、口腔唾液、肿瘤组织等
FISH探针设计、评估报告
1. 单探针效果很好,乙方提供单探针设计、项目报告。提供序列信息和探针。
Dubosiella newyorkensis 单探针特异性较好
目标物种名称: Dubosiella newyorkensis ,属于目标物种的16S序列有 2 条 (下称目标16S序列库), Dubosiella 的物种有 310 条 ,属于非目标物种的序列有 17961 88 条 (下称非目标序列库)。
单 探针在库中匹配情况统计:
p1_ 目标 mismatch0_count | p1_ 目标 mismatch1_count | p1_ 目标 mismatch2_count | p1_ 目标 mismatch3_count | p1_ 目标 mismatch0_percent | p1_ 目标 mismatch1_percent | p1_ 目标 mismatch2_percent | p1_ 目标 mismatch3_percent |
2 | 2 | 2 | 2 | 100 | 100 | 100 | 100 |
2. 单探针效果不好,乙方在设计过程中自动升级提供双探针设计、项目报告。提供序列信息和探针。
Salmonella enterica subsp. enterica serovar Typhi 单探针设计不好,双探针显著改善。
目标物种名称: Salmonella enterica subsp. enterica serovar Typhi ,属于目标物种的 16S 序列有 96 条(下称目标 16S 序列库), Salmonella enterica 的物种有 1792 条,属于非目标物种的序列有 1796094 条(下称非目标序列库)。
设计的第一条探针 在库中匹配情况统计 :
p1_ 非目标 mismatch0_count | p1_ 非目标 mismatch1_count | p1_ 非目标 mismatch2_count | p1_ 非目标 mismatch3_count | p1_start |
3453 | 6317 | 9039 | 10828 | 447 |
p1_ 目标 mismatch0_count | p1_ 目标 mismatch1_count | p1_ 目标 mismatch2_count | p1_ 目标 mismatch3_count | p1_ 目标 mismatch0_percent | p1_ 目标 mismatch1_percent | p1_ 目标 mismatch2_percent | p1_ 目标 mismatch3_percent |
92 | 93 | 93 | 93 | 95.83333 | 96.875 | 96.875 | 96.875 |
设计的第二条探针 在序列库中匹配情况统计 :
p2_ 非目标 mismatch0_count | p2_ 非目标 mismatch1_count | p2_ 非目标 mismatch2_count | p2_ 非目标 mismatch3_count | p2_start |
69282 | 85392 | 87251 | 89460 | 843 |
p2_ 目标 mismatch0_count | p2_ 目标 mismatch1_count | p2_ 目标 mismatch2_count | p2_ 目标 mismatch3_count | p2_ 目标 mismatch0_percent | p2_ 目标 mismatch1_percent | p2_ 目标 mismatch2_percent | p2_ 目标 mismatch3_ percent |
95 | 95 | 95 | 95 | 98.95833 | 98.95833 | 98.95833 | 98.95833 |
两条探针分别匹配数据库后,共同击中非目标序列库的序列条数:
p12_ 非目标 mismatch0_count | p12_ 非目标 mismatch1_count | p12_ 非目标 mismatch2_count | p12_ 非目标 mismatch3_count |
3336 | 6187 | 8893 | 10694 |
说明:
1. p*n_ 非目标 mismatch*_ count :探针 prob1 或 2 与 非目标序列库 进行匹配,分别统计 mismatch 为 0.1.2.3 时的序列条数。
2. p*_start :探针 prob1 或 2 在 16S 序列上的起始位置。
3. p*_ 目标 mismatch*_count :探针 prob1 或 2 与 目标 16S 序列库 匹配的条数,分别统计 mismatch 为 0.1.2.3 时的序列条数。
4 .p*_ 目标 mismatch*_percent :探针 prob1 或 2 与 目标 16S 序列库 匹配 序列 条数 对应 的百分比,分别统计 mismatch 为 0.1.2.3 时的序列条数的百分比。
建议:单条 FISH探针特异性较差,如需提高特异性,建议合成第二条探针(标记不同荧光),进行双FISH探针杂交检测。
文献解读 一、特异性荧光原位杂交探针的设计
鼠李糖乳杆菌 Probio-M9的特异性荧光原位杂交探针设计及应用 2021
目的 为了研究特定菌株在宿主肠中的定植和精确定位,使用荧光原位杂交定位鼠李糖乳杆菌 Probio – M9。
实验设计
1. 鼠李糖乳杆菌种水平特异性探针( Probe-16S)设计
根据现有的乳杆菌的基因组信息, 筛选鼠李糖乳杆菌基因组的特定片段,根据鼠李糖乳杆菌的 16S 序列设计了特异性探针( 5’-CGCCGACAACAGTTACTCTGCCGACC-3’), 并在5’端用6-羧基荧光素(6-FAM)荧光基团修饰。杂交优化后,使用鼠李糖乳杆菌的15个代表性菌株和乳杆菌相关属的15个代表性菌株和乳杆菌相关属的15个代表性菌株测试了探针16S的特异性。
2. 鼠李糖杆菌 Probio-M9菌株水平特异性探针(Probe-SP)设计
根据现有乳酸杆菌的基因组信息, 首先筛选鼠李糖乳杆菌基因组的特定片段,然后将鼠李糖乳杆菌不同菌株的基因组与 Probio-M9 菌株的基因组进行比较和分析,以筛选特异性基因组片段。基于该片段,设计了 Probio-M9菌株的水平特异性探针(5′-CCAACTGACGCCTTCACTTCG)。 使用鼠李糖乳杆菌的15个代表性菌株和来自乳杆菌的相关属的15个代表性菌株测试探针-SP 的特异性。
结果展示

本研究针对鼠李糖乳杆菌 Probio-M9 和其它相关物种的基因组序列 , 分别设计了种水平特异性探针( Probe-16S)和菌株水平特异性荧光原位杂交探针(Probe-SP),并通过对15株鼠李糖乳杆菌菌株和15株其它益生菌进行探针特异性的验证;同时结合荧光 D -氨基酸(FDAA)代谢探针,在大鼠的肠道中检测到鼠李糖乳杆菌Probio-M9的活细胞。本方法在在摄入鼠李糖乳杆菌Probio-M9菌株后可利用该探针检测及定位宿主肠道内的鼠李糖乳杆菌Probio-M9, 为以后开发和利用Probio-M9提供更多的理论依据。
二、 荧光原位杂交确定肿瘤内微生物的存在
Intratumoural microbiome can predict the prognosis of hepatocellular carcinoma after surgery 2023
实验目的及实验设计 使用荧光原位杂交 法测定 了肿瘤内微生物组的存在,并用 16S rDNA测序 法研究 了肿瘤和邻近正常组织中的微生物群落 分布 。对配对组的微生物特征进行了 鉴别 ,并进一步研究了它们与临床特征的相关性 。用 肝型对肿瘤组织的微生物特征进行 分 类,进一步分析 了 肝型的独立预后价值 。
部分 结果展示 肝癌中存在微生物

为了揭示 肝癌 中微生物 群 的存在,使用 UB388探针对肿瘤样本进行FISH 检测 。
(图 A )通过检测 FISH信号证实了肝细胞癌中 存在 细菌。 EUB388探针(红色)和DAPI(蓝色)染色的肿瘤样品中细菌16S rRNA的FISH分析。
(图 B ) 表明 一些 FISH信号 存在于 CD8+细胞中,表明CD8+细胞 中 存在细菌。 细菌 16S rRNA与免疫细胞共染色。CD8+T细胞用粉色(B)标记 。
(图 C ) CD68+巨噬细胞 倾向于 分布在瘤内微生物更丰富的区域, 提示 瘤内微生物组可能影响 肝癌 中巨噬细胞的浸润。 巨噬细胞用绿色 (C)标记。
肿瘤和邻近正常组织 中 不同 的 微生物 分布


对 91名 肝癌 患者的肿瘤 样本 和邻近样本进行检查。 肝细胞癌 (HCC)肿瘤和邻近正常组织中微生物组的表征。
( 图 A ) а -多样性显示肿瘤和邻近组织之间没有显着差异(p= 0. 436)。
(图 B ) β多样性揭示了肿瘤组和正常组之间的显着差异(p< 0. 001)。
(图 C ) PLS-DA揭示了两组之间的多样性。四个细菌门(变形菌门、放线菌门、厚壁菌门和奇球菌-栖热菌门)占序列的90%。
(图 D )四个门在肿瘤和邻近正常组织中均占 优势 。
本实验中使用 荧光原位杂交确定了肿瘤内微生物组的存在 ,具有特异性好 、 定位准确 、 检测时间短 , 试验周期短等的优势,更适于肿瘤样本微生物多样性的检测。

 AG亚游生物 您自己的微生态研究团队|专注微生态研究与应用
AG亚游生物 您自己的微生态研究团队|专注微生态研究与应用